Análisis de datos con Pandas
Análisis de datos con Pandas
- Pandas (cuyo nombre proviene de Panel Data) es una librería de Python diseñada para la manipulación y el análisis de datos estructurados (datos tabulados, series temporales).
- Ofrece dos estructuras de datos fundamentales: Series y DataFrame.
- Pandas está construido sobre NumPy.
- Hoy en día es una de las librerías fundamentales para la ciencia de datos junto a NumPy, SciPy, statsmodels, scikit-learn, matplotlib, etc.
- Algunos de sus principales usos
son:
- La importación y exportación de datos a diferentes formatos: CSV, Excel, JSON, HTML, Stata, SQL, etc.
- Seleccionar y filtrar datos; agregar, modificar y eliminar filas y columnas a tablas
- Manejar datos faltantes (NaNs)
- Realizar análisis estadísticos
- Agrupar datos (groupby()) y fusionar o combinar datasets (merge())
- Recursos adicionales:
Series
- Una Serie es un array 1-dim cuya secuencia de valores está asociada a un índice de etiquetas.
- Por defecto, el índice es numérico. Si la Serie tiene longitud $N$, el índice contendrá los enteros desde $0$ a $N - 1$.
- El índice nos permite seleccionar uno o varios elementos a través de sus etiquetas.
- ¡Operar sobre la Serie a través de funciones de NumPy no cambia el índice!
- isnull() nos permite comprobar si hay datos faltantes.
- ¡Y las operaciones sobre varias series (e.g. sumas) se alinean con respecto a los índices!
datos = [3, -2, 0, 42]
obj = pd.Series(datos)
print(obj)
# 0 3
# 1 -2
# 2 0
# 3 42
# dtype: int64
# Creamos una serie especificanado un índice
obj2 = pd.Series(datos, index=['a', 'd', 'g', 'w'])
print(obj2)
# a 3
# d -2
# g 0
# w 42
# dtype: int64
obj2 = pd.Series(datos, index=['a', 'd', 'g', 'w'])
print(obj2)
# a 3
# d -2
# g 0
# w 42
# dtype: int64
# Selección de datos a través de etiquetas
print(obj2['a']) # 3
obj2['w'] = 100
print(obj2[['a', 'w']])
# a 3
# w 100
# dtype: int64
# El filtrado y las operaciones mantienen las etiquetas
print(obj2[obj2 > 70])
# w 100
# dtype: int64
print(obj2 * 3)
# a 9
# d -6
# g 0
# w 300
# dtype: int64
print(np.exp(obj2))
# a 2.008554e+01
# d 1.353353e-01
# g 1.000000e+00
# w 2.688117e+43
# dtype: float64
# Creamos una serie a partir de un diccionario
datos_ciudades = {'A Coruña': 249261, 'Lugo': 99482,
'Ourense': 104891, 'Pontevedra': 83077}
# Pero damos un índice distinto al del diccionario
ciudades = pd.Series(datos_ciudades,
index=['Santiago', 'Lugo', 'Ourense', 'Vigo'])
print(ciudades)
# Santiago NaN
# Lugo 99482.0
# Ourense 104891.0
# Vigo NaN
# dtype: float64
pd.isnull(ciudades) # También existe notnull()
# Santiago True
# Lugo False
# Ourense False
# Vigo True
# dtype: bool
print(ciudades)
# Santiago NaN
# Lugo 99482.0
# Ourense 104891.0
# Vigo NaN
# dtype: float64
pd.isnull(ciudades) # También existe notnull()
# Santiago True
# Lugo False
# Ourense False
# Vigo True
# dtype: bool
nuevos_nacimientos = pd.Series({'Ourense': 5000, 'Vigo': 5000})
print(ciudades + nuevos_nacimientos)
# Lugo NaN
# Ourense 109891.0
# Santiago NaN
# Vigo NaN
# dtype: float64
DataFrames
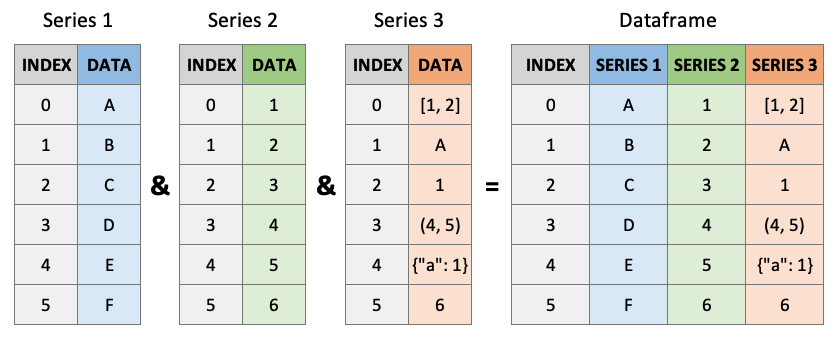
DataFrames

DataFrames
- Un DataFrame representa una tabla de datos y contiene una colección ordenada de columnas, que pueden tener distintos tipos de datos.
- Un DataFrame tiene dos índices: el de filas y el de columnas.
- Existen diferentes formas de crear un DataFrame.
Desde un diccionario de listas/arrays de NumPy
datos = {
'nombre': ["Marcos", "Eva", "Xoel"],
'edad': [40, 35, 23],
'tiene_perro': [False, True, True]}
# columns permite reordenar las columnas
# Si no se encuentran los datos para
# alguna columna, sus valores son NaNs
df=pd.DataFrame(datos, columns=['nombre',
'tiene_perro', 'sueldo', 'edad'])
print(df)
# nombre tiene_perro sueldo edad
# 0 Marcos False NaN 40
# 1 Eva True NaN 35
# 2 Xoel True NaN 23
Desde una lista/array de diccionarios
datos2 = [
{'nombre': "Marcos", 'edad': 40,
'tiene_perro': False},
{'nombre': "Eva", 'edad': 35,
'tiene_perro': True},
{'nombre': "Xoel", 'edad': 23,
'tiene_perro': True}
]
df2 = pd.DataFrame(datos2)
print(df2)
# nombre edad tiene_perro
# 0 Marcos 40 False
# 1 Eva 35 True
# 2 Xoel 23 True
Desde archivos
(CSV, Excel, JSON...)
# Desde un archivo CSV
df3=pd.read_csv('ruta/al/archivo.csv',
header=0, # header en la primera línea
sep="\t") # campos separados por tabs
# Desde un archivo Excel
df4=pd.read_excel('ruta/al/archivo.xlsx')
# Desde un archivo JSON
df5=pd.read_json('ruta/al/archivo.json')
Nota 1: Hay más posibilidades, como a partir de nested
dictionaries.
Nota 2: Para mostrar un DataFrame, es preferible usar df.head(n) a
print(df).
Nota 3: df.info() es muy útil para comprobar, entre otros, el tipo de datos de cada columna y el número
de filas y columnas.
Selección de datos
Podemos seleccionar columnas específicas a través
de su nombre,
con dos posibilidades:
columna_nombre = df['nombre']
columna_nombre = df.nombre # Mismo resultado que df['nombre']
print(columna_nombre)
# 0 Marcos
# 1 Eva
# 2 Xoel
# Name: nombre, dtype: object
print(type(columna_nombre)) # class 'pandas.core.series.Series'
Y también podemos seleccionar varias columnas a la vez:
filtrado = df[['nombre', 'edad']]
print(filtrado)
# nombre edad
# 0 Marcos 40
# 1 Eva 35
# 2 Xoel 23
Selección de datos
Para seleccionar filas y filtrar según una condición, podemos usar la misma notación que con series:
print(df[:2])
# nombre edad tiene_perro
# 0 Marcos 40 False
# 1 Eva 35 True
# "and" y "or" no funcionan, como en NumPy
filtrado = df[(df['edad'] < 30) | (df['nombre'] == "Marcos")]
print(filtrado)
# nombre edad tiene_perro
# 0 Marcos 40 False
# 2 Xoel 23 True
Selección de datos
Para seleccionar por filas y columnas a la vez, se puede usar loc[], al que se le deben pasar etiquetas, o iloc[], al que se le debe pasar la posición de la fila/columna como un entero.
print(df.loc[:, 'tiene_perro']) # Todas las filas, columna 'tiene_perro'
# 0 False
# 1 True
# 2 True
# Name: tiene_perro, dtype: bool
print(df.iloc[1:, 1]) # Filas de 1 en adelante, primera columna
# 1 Eva
# 2 Xoel
# Name: nombre, dtype: object
print(df.iloc[1:, 1][df['edad'] < 28]) # Filtrado después de indexar
# 2 23
# Name: edad, dtype: int64
Operaciones aritméticas con DataFrames
Muy similares a las de las series, pero alineando los datos tanto por fila como por columna.
df1 = pd.DataFrame(np.arange(12).reshape((4, 3)), columns=['c1', 'c2', 'c3'], index=['r1', 'r2', 'r3', 'r4'])
df2 = pd.DataFrame(np.full((4, 4), 100), columns=['c1', 'c2', 'c4', 'c5'], index=['r1', 'r2', 'r4', 'r5'])
print(df1)
# c1 c2 c3
# r1 0 1 2
# r2 3 4 5
# r3 6 7 8
# r4 9 10 11
print(df2)
# c1 c2 c4 c5
# r1 100 100 100 100
# r2 100 100 100 100
# r4 100 100 100 100
# r5 100 100 100 100
print(df1 + df2)
# c1 c2 c3 c4 c5
# r1 100.0 101.0 NaN NaN NaN
# r2 103.0 104.0 NaN NaN NaN
# r3 NaN NaN NaN NaN NaN
# r4 109.0 110.0 NaN NaN NaN
# r5 NaN NaN NaN NaN NaN
Operaciones de reshaping
- Ordenar las filas en función de los valores de una columna (por defecto, orden ascendente, i.e., ascending=True): df.sort_values('col1', ascending=False)
- Ordenar las filas en función de los valores de varias columnas (por defecto, orden ascendente, i.e., ascending=True): df.sort_values(['col1', 'col2'], ascending=False)
- Ordenar las filas en función del índice (por defecto, orden ascendente, i.e., ascending=True): df.sort_index(), df.sort_index(ascending=False)
- Construir un nuevo índice donde las etiquetas enumeran las filas:
df.reset_index().
Nota: el antiguo índice pasa a ser una columna. - Construir un nuevo índice donde las etiquetas enumeran las filas y eliminar el antiguo índice: df.reset_index(drop=True)
- Añadir una nueva columna con nombre 'nueva_col': df['nueva_col'] = ...
- Borrar columnas: df.drop(columns=['col1', 'col2'])
- Borrar filas a partir de su etiqueta en el índice (en este ejemplo, las filas de etiquetas 0 y 1): df.drop([0, 1])
Estas operaciones, al igual que la mayoría de métodos de Pandas, devuelven DataFrames. Por tanto, pueden encadenarse, e.g. df.sort_values('col1').reset_index(drop=True)
# Crear un DataFrame de ejemplo
datos = {
'col1': [3, 1, 4, 1, 5],
'col2': [9, 2, 6, 5, 3],
'col3': ['a', 'b', 'c', 'd', 'e']
}
df = pd.DataFrame(datos)
print(df)
# col1 col2 col3
# 0 3 9 a
# 1 1 2 b
# 2 4 6 c
# 3 1 5 d
# 4 5 3 e
# Ordenar por col1 y col2 en orden ascendente
df_sorted = df.sort_values(['col1', 'col2'])
print(df_sorted)
# col1 col2 col3
# 1 1 2 b
# 3 1 5 d
# 0 3 9 a
# 2 4 6 c
# 4 5 3 e
# Ordenar por índice en orden descendente
df_sorted_index = df.sort_index(ascending=False)
print(df_sorted_index)
# col1 col2 col3
# 4 5 3 e
# 3 1 5 d
# 2 4 6 c
# 1 1 2 b
# 0 3 9 a
# Resetear el índice
df_reset = df.reset_index()
print(df_reset)
# index col1 col2 col3
# 0 0 3 9 a
# 1 1 1 2 b
# 2 2 4 6 c
# 3 3 1 5 d
# 4 4 5 3 e
# Añadir una nueva columna
df['nueva_col'] = df['col1'] + df['col2']
print(df)
# col1 col2 col3 nueva_col
# 0 3 9 a 12
# 1 1 2 b 3
# 2 4 6 c 10
# 3 1 5 d 6
# 4 5 3 e 8
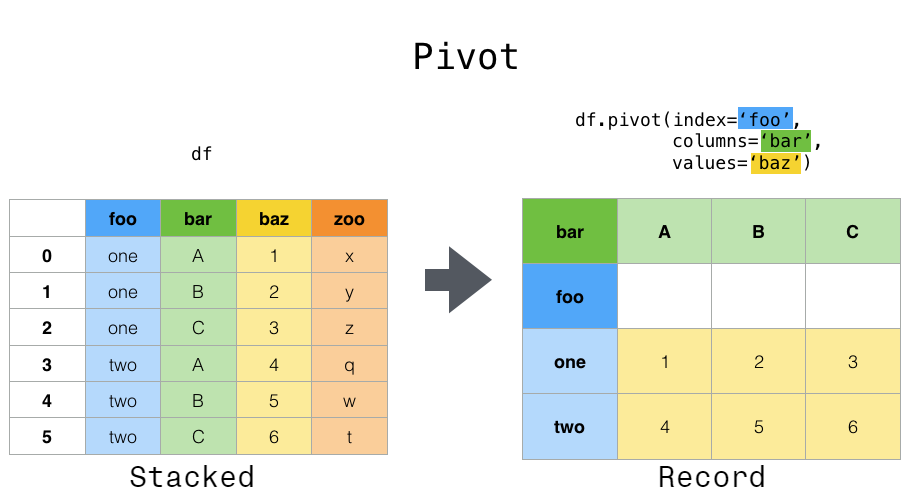
Operaciones de reshaping
df.pivot() convierte los datos de formato largo a ancho.
df.melt() convierte los datos de formato ancho a largo.
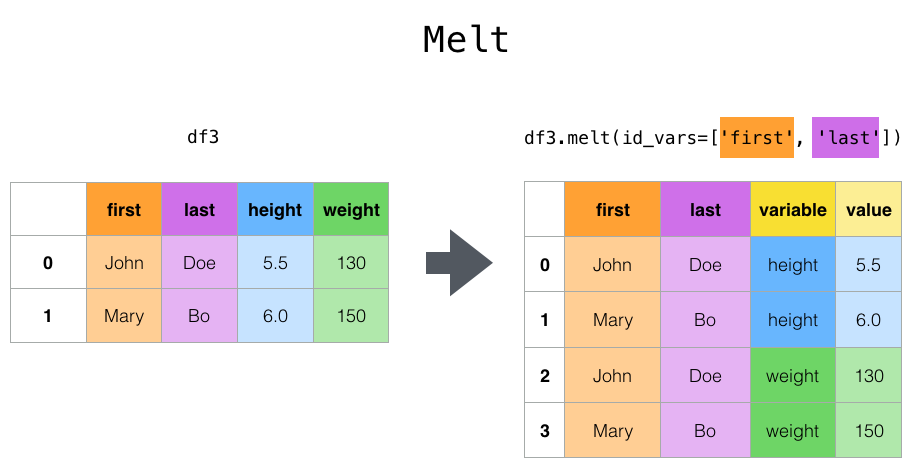
Transformación de datos
La función apply() permite aplicar una función a lo largo de un eje de un DataFrame o de los elementos de una serie.
- axis=0: aplica la función a cada columna. Es la opción por defecto.
- axis=1: aplica la función a cada fila.
df = pd.DataFrame({
'producto': ['A', 'B', 'C'],
'precio': [10, 20, 30]
})
df['precio_ajustado'] = df.apply(lambda row: row['precio'] * 1.10, axis=1)
print(df)
# producto precio precio_ajustado
# 0 A 10 11.0
# 1 B 20 22.0
# 2 C 30 33.0
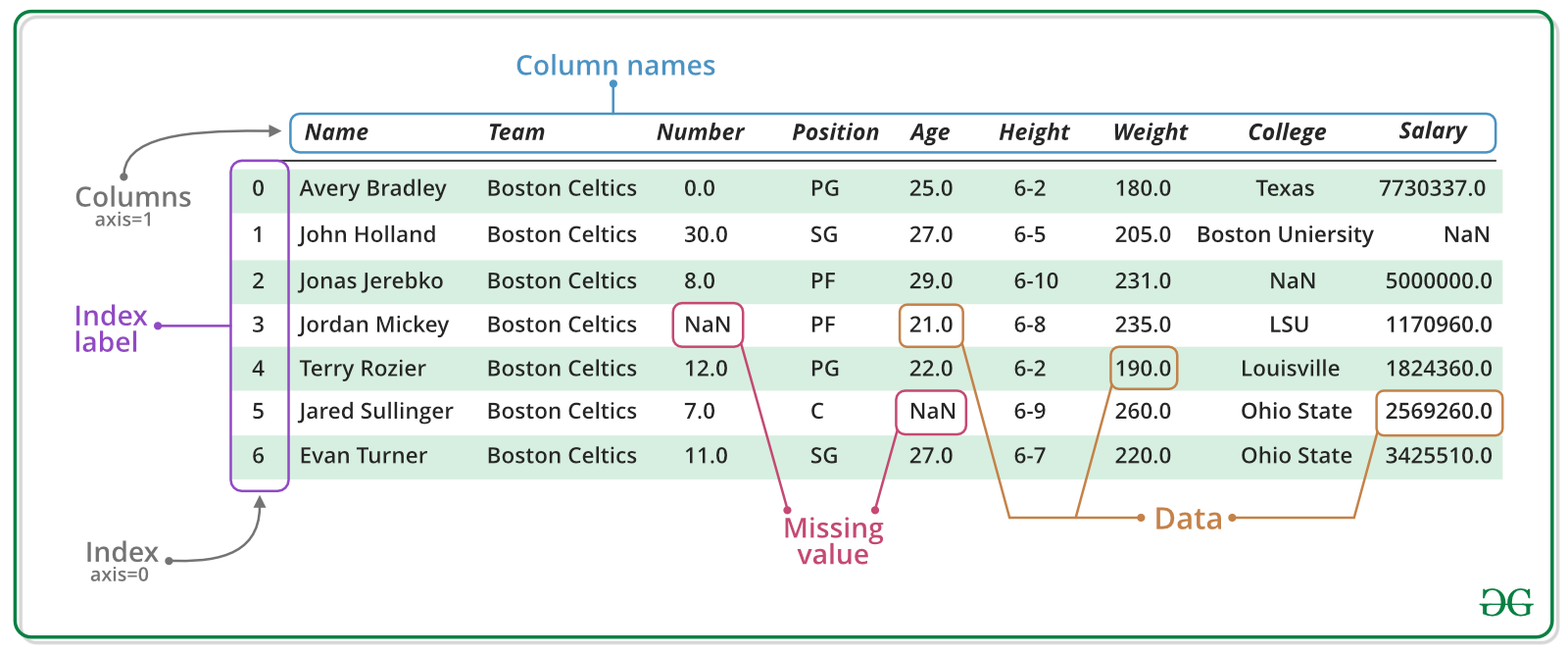
Limpieza de datos
En los proyectos de análisis de datos es muy común encontrar datos faltantes (NaNs), errores de medición, datos duplicados, etc. Es importante saber limpiarlos de forma adecuada.Existen varios métodos de utilidad:
- dropna(): Elimina todas las filas y columnas que contienen NAs
datos = pd.DataFrame([[1., 6.5, 2.],
[1., np.nan, np.nan],
[np.nan, np.nan, np.nan],
[np.nan, 6.5, 3.]])
datos_limpios = datos.dropna()
print(datos)
# 0 1 2
# 0 1.0 6.5 2.0
# 1 1.0 NaN NaN
# 2 NaN NaN NaN
# 3 NaN 6.5 3.0
print(datos_limpios)
# 0 1 2
# 0 1.0 6.5 2.0
- dropna(how="all"): Elimina solo las filas en las que todos los datos son NAs
- dropna(how="all", axis=1): Elimina solo las columnas en las que todos los datos son NAs
datos = pd.DataFrame([[1., 6.5, 2.],
[1., np.nan, np.nan],
[np.nan, np.nan, np.nan],
[np.nan, 6.5, 3.]])
datos_limpios = datos.dropna(how="all")
print(datos)
# 0 1 2
# 0 1.0 6.5 2.0
# 1 1.0 NaN NaN
# 2 NaN NaN NaN
# 3 NaN 6.5 3.0
print(datos_limpios)
# 0 1 2
# 0 1.0 6.5 2.0
# 1 1.0 NaN NaN
# 3 NaN 6.5 3.0
Es común usar DataFrames para guardar datos recogidos a lo largo del tiempo. Cada columna corresponde a los atributos de interés y cada fila, a los datos recogidos en un instante de tiempo. En tal caso, se puede elegir eliminar aquellas filas que no tienen un número de mediciones suficientes.
- dropna(thresh=n): Elimina las filas que no tienen al menos $n$ valores no NAs
datos = pd.DataFrame([[1., 6.5, 2.], [1., np.nan, np.nan], [np.nan, np.nan, np.nan], [np.nan, 6.5, 3.]])
datos_limpios = datos.dropna(thresh=2)
print(datos)
# 0 1 2
# 0 1.0 6.5 2.0
# 1 1.0 NaN NaN
# 2 NaN NaN NaN
# 3 NaN 6.5 3.0
print(datos_limpios)
# 0 1 2
# 0 1.0 6.5 2.0
# 3 NaN 6.5 3.0
En lugar de eliminar los valores faltantes, podemos optar por rellenar sus posiciones con un determinado valor.
- fillna(): Sustituye los NAs por el valor pasado como argumento.
datos = pd.DataFrame([[1., 6.5, 2.], [1., np.nan, np.nan], [np.nan, np.nan, np.nan], [np.nan, 6.5, 3.]])
datos_limpios = datos.fillna(0)
datos_limpios_2 = datos.fillna(datos.mean()) # También admite otras estructuras de datos
print(datos)
# 0 1 2
# 0 1.0 6.5 2.0
# 1 1.0 NaN NaN
# 2 NaN NaN NaN
# 3 NaN 6.5 3.0
print(datos_limpios); print(datos_limpios_2)
# 0 1 2 0 1 2
# 0 1.0 6.5 2.0 0 1.0 6.5 2.0
# 1 1.0 0.0 0.0 1 1.0 6.5 2.5
# 2 0.0 0.0 0.0 2 1.0 6.5 2.5
# 3 0.0 6.5 3.0 3 1.0 6.5 3.0
También es importante localizar y eliminar filas duplicadas cuando tiene sentido hacerlo.
- duplicated(): Devuelve una serie indicando, para cada fila, si está duplicada o no.
- drop_duplicates(): Elimina las filas duplicadas.
- A ambos métodos se les pueden indicar solo columnas concretas.
df = pd.DataFrame({'c1': ['a'] * 3 + ['b'] * 2,
'c2': [1, 1, 2, 3, 3]})
print(df); print(df.duplicated())
# c1 c2
# 0 a 1 0 False
# 1 a 1 1 True
# 2 a 2 2 False
# 3 b 3 3 False
# 4 b 3 4 True
# dtype: bool
print(df.drop_duplicates()); print(df.drop_duplicates().reset_index(drop=True))
# c1 c2 c1 c2
# 0 a 1 0 a 1
# 2 a 2 1 a 2
# 3 b 3 2 b 3
Los outliers pueden deberse a errores de medición o de inserción en la bases de datos. En estos casos, es recomendable eliminarlos.
Hay diferentes opciones: eliminarlos, sustituirlos por el máximo/por la media, etc.
datos_ph = pd.DataFrame(16 * np.random.rand(1000, 4), columns=["detector1", "detector2", "detector3", "detector4"])
print(datos_ph.describe())
# detector1 detector2 detector3 detector4
# count 1000.000000 1000.000000 1000.000000 1000.000000
# mean 8.008337 8.267079 7.916209 8.024089
# std 4.722665 4.585955 4.644795 4.587050
# min 0.023579 0.070377 0.020743 0.004057
# 25% 3.812108 4.474951 3.877351 4.281892
# 50% 8.007732 8.042149 7.647697 7.886486
# 75% 12.133669 12.261288 12.005321 11.885338
# max 15.988374 15.999366 15.986327 15.979152
datos_ph[datos_ph > 14] = 14
print(datos_ph.describe())
# detector1 detector2 detector3 detector4
# count 1000.000000 1000.000000 1000.000000 1000.000000
# mean 7.875772 7.881265 7.894457 7.947086
# std 4.457306 4.422518 4.379599 4.390199
# min 0.036536 0.026266 0.001390 0.008563
# 25% 3.836448 4.154702 4.154869 4.094805
# 50% 8.132162 7.936885 7.928288 7.960573
# 75% 12.143415 12.058752 11.999224 12.138702
# max 14.000000 14.000000 14.000000 14.000000
Análisis estadísticos
- Resumen de los datos numéricos (media, std, min, max, percentiles): describe()
- Medidas de centralización: mean(), median(), mode()
- Medidas de dispersión: std(), var(), mad()
- Medidas de posición relativa: quantile(q), min(), max()
- Otras: sum(), count() - cuenta el número de valores no nulos, value_counts() - cuenta el número de filas distintas, etc.
- corr() calcula la correlación entre columnas (de Pearson, Spearman o Kendall)
- cov() calcula la matriz de covarianzas
Otras librerías, como scipy, permiten realizar operaciones más avanzadas, como contrastes de hipótesis.
df = pd.DataFrame({
"A": [1, 2, 3, 4, 5],
"B": [2, 4, 6, 8, 10],
"C": [np.nan, 3, 8, 6, 7]
})
print(df.describe())
# A B C
# count 5.000000 5.000000 4.000000
# mean 3.000000 6.000000 6.000000
# std 1.581139 3.162278 2.160247
# min 1.000000 2.000000 3.000000
# 25% 2.000000 4.000000 5.250000
# 50% 3.000000 6.000000 6.500000
# 75% 4.000000 8.000000 7.250000
# max 5.000000 10.000000 8.000000
print(df.corr())
# A B C
# A 1.000000 1.000000 0.597614
# B 1.000000 1.000000 0.597614
# C 0.597614 0.597614 1.000000
Combinación de datasets
En muchas ocasiones, los datos de un proyecto están en archivos o formatos distintos. Para poder utilizarlos, es conveniente combinarlos en una única estructura de datos.- merge(): Combinación de filas de datasets en base a una o más columnas comunes (producto cartesiano).
- Por defecto, merge() los combina en base a las
columnas que tienen el mismo nombre.
Se pueden indicar explícitamente
con el argumento on.
Ejemplo: pd.merge(df1, df2, on=["col_comun_1", "col_comun_2"]) - Si la columna común tiene un nombre distinto en cada dataframe, se pueden referenciar por separado: pd.merge(df_left, df_right, left_on="left_col", right_on="right_col")
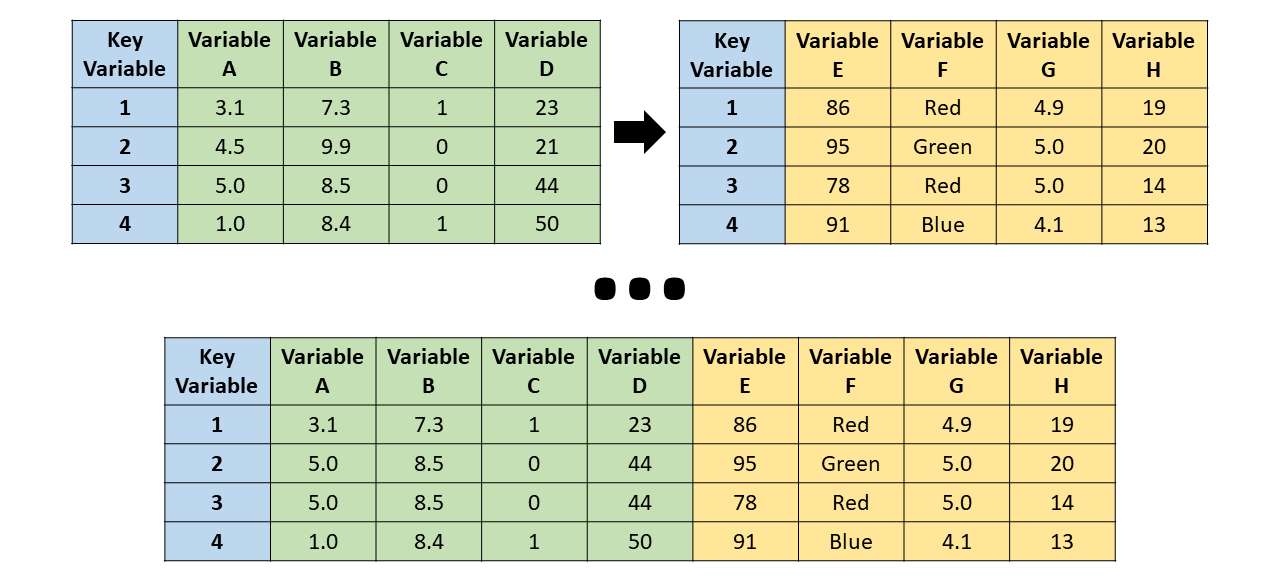
- merge(): Combinación de filas de datasets en base a una o más columnas comunes (producto cartesiano).
- Por defecto, merge() los combina en base a las
columnas que tienen el mismo nombre.
Se pueden indicar explícitamente
con el argumento on.
Ejemplo: pd.merge(df1, df2, on=["col_comun_1", "col_comun_2"]) - Si la columna común tiene un nombre distinto en cada dataframe, se pueden referenciar por separado: pd.merge(df_left, df_right, left_on="left_col", right_on="right_col")
df1 = pd.DataFrame({"frutas": ["manzanas", "platanos", "limones", "uvas", "manzanas"],
"unidades": np.random.randint(1, 10, 5)}) # 5 enteros aleatorios en el intervalo [1, 10)
df2 = pd.DataFrame({"frutas": ["manzanas", "platanos", "limones", "uvas"],
"precio": np.random.random(4) * 10}) # (4 floats aleatorios en el intervalo [0, 1)) * 10
print(pd.merge(df1, df2))
# frutas unidades precio
# 0 manzanas 5 5.630322
# 1 manzanas 4 5.630322
# 2 platanos 7 4.570918
# 3 limones 6 3.348530
# 4 uvas 8 6.413218
# Equivalentes: pd.merge(df1, df2, on="frutas"), pd.merge(df1, df2, left_on="frutas", right_on="frutas")
- ¿Qué pasa si alguno de los DataFrames no tiene datos para todos los valores de la columna común?
- Por defecto, en el resultado de merge() solo aparecerán aquellos valores que están en ambos DataFrames. Es decir, se hace una intersección sobre la columna ("inner join").
- Con el parámetro how podemos controlar este comportamiento.
- how="outer": toma la unión de los valores y rellena con NaNs.
- how="left": toma solo los valores de la columna del primer DataFrame. Si alguno no aparece en el segundo DataFrame, rellena con NaNs. how="right" es análogo.
df1 = pd.DataFrame({"frutas": ["manzanas", "platanos", "limones"],
"unidades": np.random.randint(1, 10, 3)}) # 3 enteros aleatorios en el intervalo [1, 10)
df2 = pd.DataFrame({"frutas": ["manzanas", "platanos", "uvas"],
"precio": np.random.random(3) * 10}) # (3 floats aleatorios en el intervalo [0, 1)) * 10
print(pd.merge(df1, df2))
# frutas unidades precio
# 0 manzanas 4 6.482856
# 1 platanos 1 1.942549
print(pd.merge(df1, df2, how="outer"))
# frutas unidades precio
# 0 manzanas 1.0 3.814989
# 1 platanos 8.0 1.374147
# 2 limones 9.0 NaN
# 3 uvas NaN 7.856911
- concat(): Concatena dos DataFrames a lo largo de las filas (axis = 0) o de las columnas (axis=1).
- Lo más común es querer descartar los índices originales. Para ello, hay que usar indicar ignore_index=True como parámetro.
- Nota: NumPy tiene una función análoga para concatenar arrays.
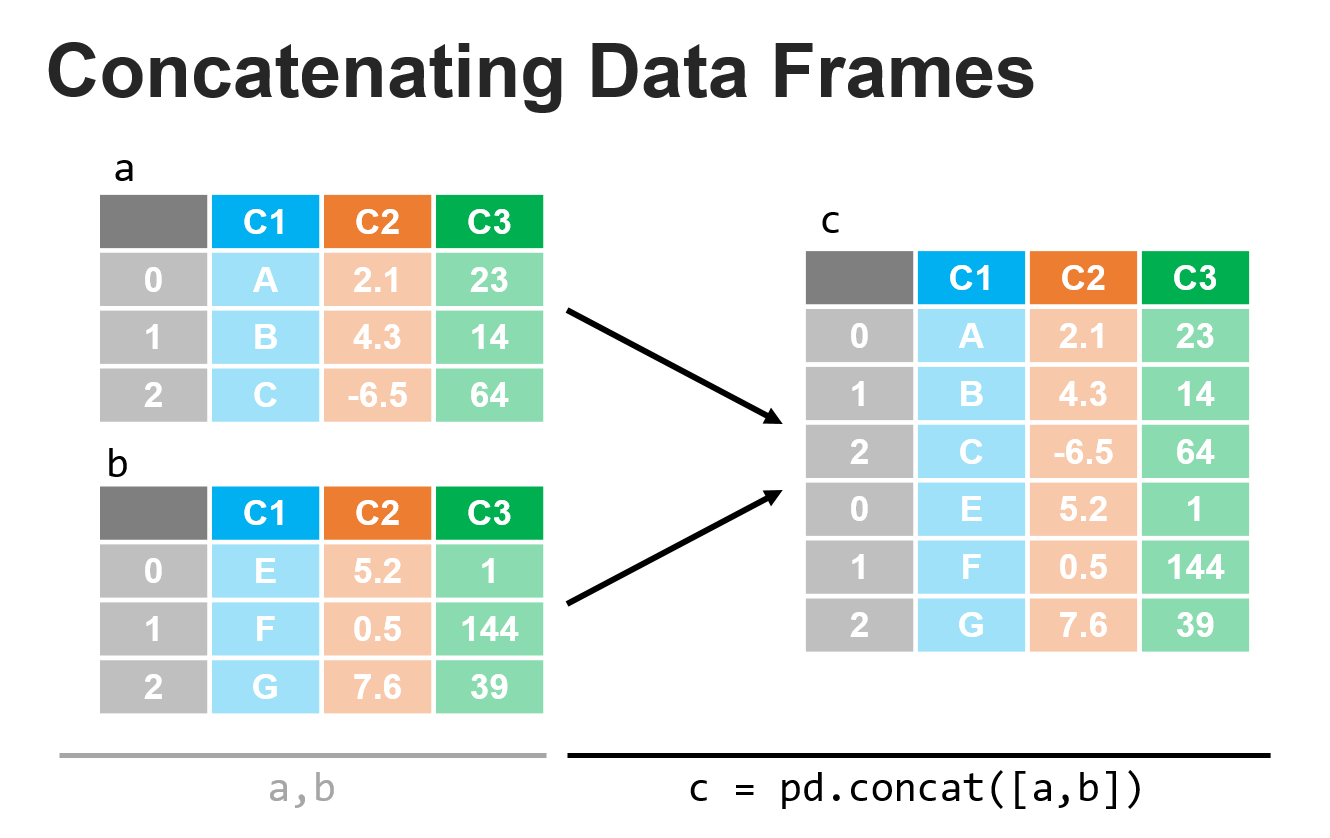
- concat(): Concatena dos DataFrames a lo largo de las filas (axis = 0) o de las columnas (axis=1).
- Lo más común es querer descartar los índices originales. Para ello, hay que usar indicar ignore_index=True como parámetro.
- Nota: NumPy tiene una función análoga para concatenar arrays.
df1 = pd.DataFrame({
"A": ["A0", "A1", "A2", "A3"],
"B": ["B0", "B1", "B2", "B3"]})
df2 = pd.DataFrame({
"A": ["A4", "A5", "A6", "A7"],
"B": ["B4", "B5", "B6", "B7"]})
print(pd.concat([df1, df2])); print(pd.concat([df1, df2], ignore_index=True))
# A B A B
# 0 A0 B0 0 A0 B0
# 1 A1 B1 1 A1 B1
# 2 A2 B2 2 A2 B2
# 3 A3 B3 3 A3 B3
# 0 A4 B4 4 A4 B4
# 1 A5 B5 5 A5 B5
# 2 A6 B6 6 A6 B6
# 3 A7 B7 7 A7 B7
Agrupación de datos
La operación de groupby() permite agrupar los datos en función a valores comunes de una variable y realizar alguna operación sobre ellos: sumarlos, tomar el máximo/mínimo, hacer la media, etc.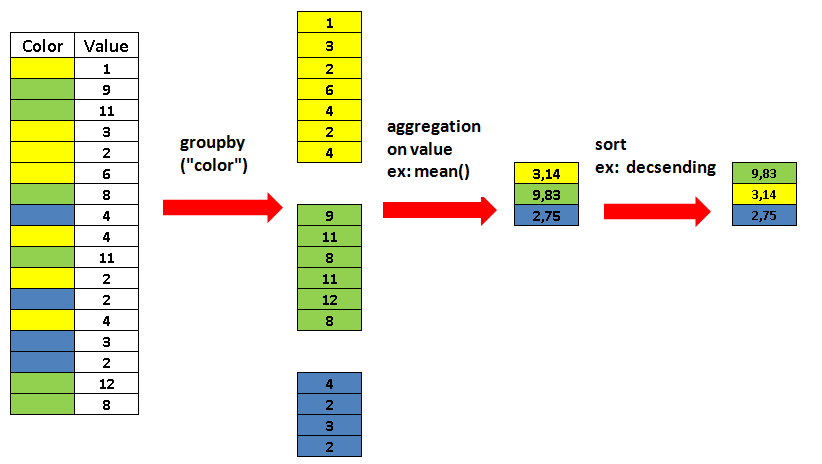
df = pd.DataFrame({
"estudiante": ["Laura", "David", "Víctor", "Laura", "David", "Víctor"],
"asignatura": ["ANM", "EDO", "TGS", "ANM", "TGS", "EDO"],
"nota": [0.5, 6.0, 9.2, 1.1, 5.0, 10]
})
print(df)
# estudiante asignatura nota
# 0 Laura ANM 0.5
# 1 David EDO 6.0
# 2 Víctor TGS 9.2
# 3 Laura ANM 1.1
# 4 David TGS 5.0
# 5 Víctor EDO 10.0
print(df[["estudiante", "nota"]].groupby("estudiante").mean())
# nota
# estudiante
# David 5.5
# Laura 0.8
# Víctor 9.6
¡Hora de practicar!
Para fijar ideas, exploraremos el siguiente dataset conteniendo información de las $1000$ películas de la historia según IMDB.